ViSeCut
ViSeCut ist eine medizinische open-source Computertomographie Bildbetrachtungs-, Bildbearbeitungs- und Bildsegmentierungssoftware, die zwei-dimensionale Computertomographie (CT) Schnittbilder (in DICOM-Format) durch verschiedene Bildverarbeitungsprozesse, Methoden und Algorithmen (volume rendering, watershed segmentation und region-growing segmentation) verarbeitet, analysiert und gewünschte Ergebnisse darstellt. ViSeCut ist unter Linux (Fedora 10) mit Programmierungssprache C++ implementiert und kann später durch Integrierung von bestimmten medizinische open-source/cross-platform Bibliotheken auch unter Windows, Mac OS X, usw. benutzt und weiter erweitert werden.
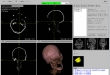
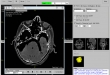
Ziel Das Ziel dieses Projektes war ein open-source Computerprogramm zu implementieren, dass im ersten Schritt als DICOM formatierte 2D-CT Schnittbilder einliest, sie dann als 2D in axial-, sagittal-, koronal Ansicht und in multiplanaren Reformation darstellt. Im zweiten Schritt sollten die geladene Bilder in einzelnen Ansichten in verschiedenen Fenstern betrachtet und bearbeitet werden, wie ein- und auszoomen, Positionsänderung einer Ansicht im Fenster, Fensterung über Hounsfield Skala, Blättern, Opazitätsänderung, Farbänderung, Punkt-Selektion, Screenshot, Bilder drehen, bestimmte Informationen vom Patient und CT-Bilder auflisten wie Patient-Name, Bilderanzahl, Bildergröße usw.
Im dritten Schritt sollten die dargestellte 2D-Bilder in einem größeren Fenster in Cine-Modus angezeigt und durch implementierte Cine-Modus Methode vorwärts/rückwärts für alle Ansichten automatisch blättert werden. Der Cine-Modus sollte auch für vergrößerte Darstellungen von 2D- und 3D-Modell angewendet werden.
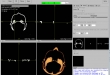
Im nächsten Schritt sollten 2D-Bilder durch bestimmte Algorithmen (volume rendering, watershed segmentation und region-growing segmentation) in einem 3D-Objekt/Modell umgewandelt werden. Vor der Segmentierung sollte das Programm in der Lage sein, dass der Benutzer sich über 2D-Bilder einen Bereich auswählt und danach Bilder segmentiert, somit sollte die Bearbeitungszeit des Algorithmus verkürzt und Zeit erspart werden.
Schließlich sollten segmentierte 3D-Modell in vtk-Format als eine Datei gespeichert werden, damit sie in einem anderen medizinischen Computerprogramm weiterverarbeitet oder betrachtet wird.
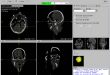
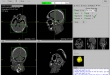
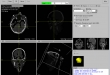
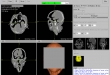
Screenshots
Features
- Einfache und Benutzerfreundliche Bedienung
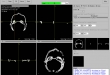
- Visualisierung der eingelesene 2D-Bilder in axial-, sagittal, koronal, Muliplanar- und 3D-Ansicht (DICOM-Darstellung)
- Bildbetrachtung für 2D- Bilder durch Maus-Knöpfe: ein- und auszoomen, Bildpositionierung aller Ansichten im separaten Fenstern, Fensterung über Hounsfield Skala, 3D-Modell drehen, Punkt Selektion über 2D-Bilder damit alle Ansichten gleiches 2D-Schnittbild anzeigt, Bereich-Selektion über 2D-Bilder für die Schneidefunktion, Blättern
- Bildbearbeitung für 2D- Bilder: Opazitätsänderung
- Bildbearbeitung für 3D-Modell: drehen, Farbeändern
- Auflisten bestimmten Bild- und Patienteninformationen
- Manuelle Eingabe bestimmter Werte für Hounsfield skala, marching cubes, volume rendering, watershed segmentation und region-growing segmentation
- Orientierung mit Beschriftungen und durch ein 3D-Modell bei der Betrachtung von 2D-Bilder und 3D-Modellen
- Cine-Modus für automatisches vorwärts/rückwärts Blättern von 2D-Serienbilder
- Vergrößerte Darstellung aller Ansichten in einem separaten Fenster
- Screenshot aller Ansichten
- Extrahieren des3D-Modelles als vtk-data
- Mitlaufender Logfunktion für durchgeführte Benutzeraktionen und Programmergebnisse
- Hilfefunktion
- Einlesen der CT-Bildern mit verschiedenen Abmessungen (512×512, 256×256 usw.)
- Vorschaufunktion bevor Laden der CT-Bilder
Y. Özbek, W. Freysinger
16.12.2011